Rosetta @ home este un proiect de calcul distribuit pentru predictia structurii proteice pe platforma Berkeley Open Infrastructure for Network Computing (BOINC), condusa de laboratorul Baker de la Universitatea din Washington.
Rosetta @ home isi propune sa prezica imbogatirea proteinelor si proiectarea de noi proteine cu ajutorul a aproximativ saizeci de mii de computere active voluntare, care proceseaza la peste 210 teraFLOPS, in medie, din 29 iulie 2016. Rosetta @ Home, isi propune sa atinga aceste obiective printr-o abordare de tip crowdsourcing.
Desi o mare parte din proiect este orientata catre cercetarea de baza pentru a imbunatati acuratetea si robustetea metodelor proteomice, Rosetta @ home face, de asemenea, cercetari aplicate asupra malariei, bolii Alzheimer si a altor patologii.
Ca toate proiectele BOINC, Rosetta @ home foloseste resurse de procesare inactiva de la calculatoarele voluntarilor pentru a efectua calcule pe unitatile de lucru individuale. Rezultatele completate sunt trimise catre un server de proiect central unde sunt validate si asimilate in bazele de date ale proiectului.
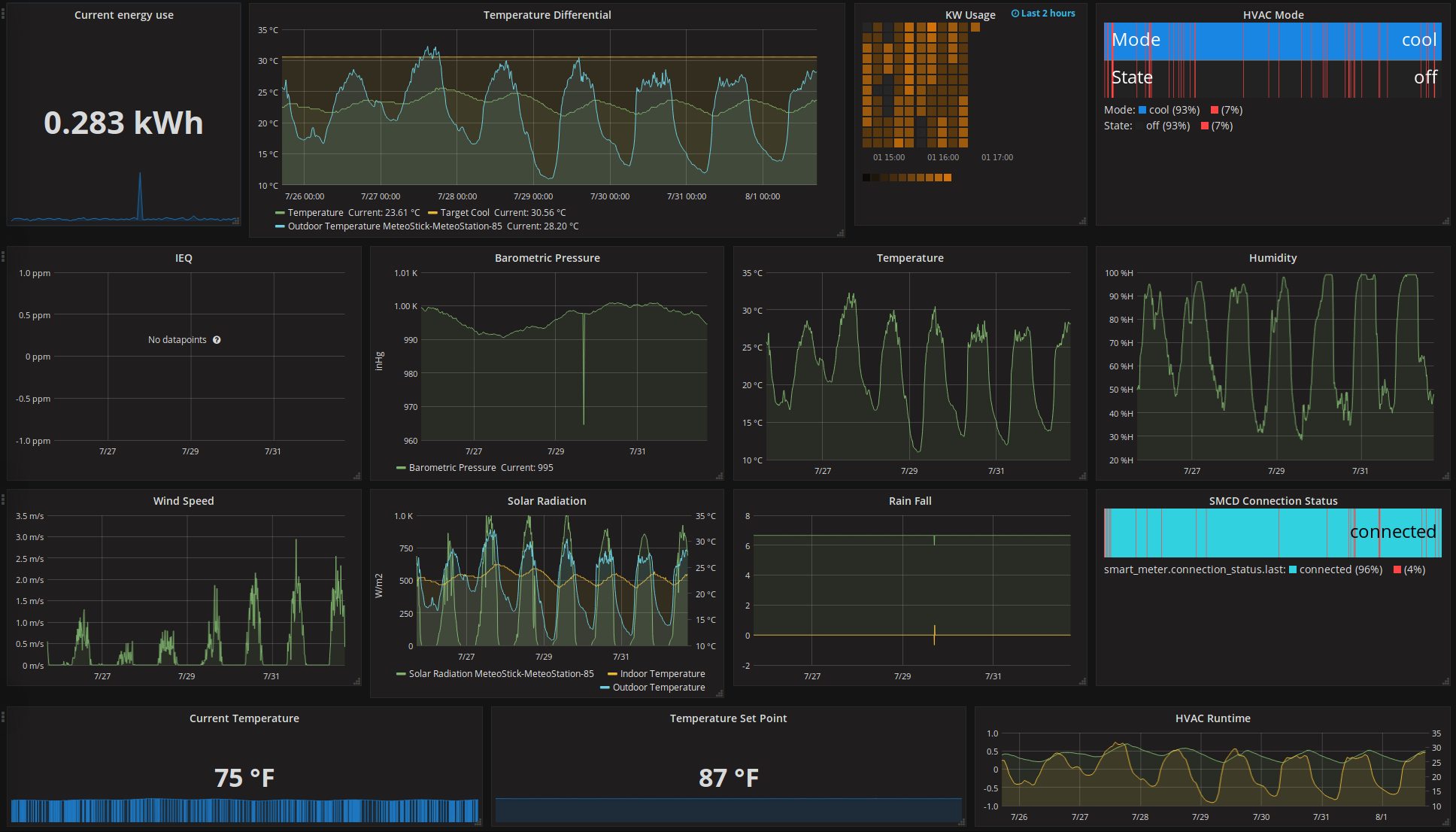
Proiectul este de tip multipla platforma si ruleaza pe o mare varietate de configuratii hardware. Utilizatorii pot vizualiza progresul structurii proteice individuale pe screensaver-ul Rosetta @ home.
Pe langa cercetarile legate de boli, reteaua Rosetta @ home serveste ca un cadru de testare a noilor metode in bioinformatica structurala. Astfel de metode sunt apoi utilizate in alte aplicatii bazate pe Rosetta, cum ar fi RosettaDock si Proiectul de pliere a proteomelor umane, dupa ce au fost suficient de dezvoltate si dovedite stabile, pe setul mare si divers de computere voluntare Rosetta @ home.
Doua teste deosebit de importante pentru noile metode dezvoltate in Rosetta @ home sunt Evaluarea critica a tehnicilor pentru prezicerea structurii proteice (CASP) si evaluarea critica a predictiilor interactiunilor (CAPRI), experimente bienale care evalueaza stadiul tehnicii in structura proteinelor. Rosetta @ home se situeaza in mod constant printre cei mai importanti prognoseri de andocare si este unul dintre cei mai buni predictori de structura tertiara disponibili.
Platforma de calcul
Aplicatia Rosetta @ home si platforma de calcul distribuit BOINC sunt disponibile pentru sistemele de operare Windows, Linux si macOS; BOINC ruleaza, de asemenea, pe cateva altele, de exemplu, FreeBSD. Participarea la Rosetta @ home necesita o unitate de procesare centrala (CPU), cu o viteza de ceas de cel putin 500 MHz, 200 megabytes de spatiu pe disc liber, 512 megabytes de memorie fizica si conectivitate la Internet.
De la 20 iulie 2016, versiunea actuala a aplicatiei Rosetta Mini este 3,73. Versiunea actuala recomandata a programului BOINC este 7.6.22. Protocolul de transfer de hipertext standard (HTTP) (port 80) este utilizat pentru comunicarea intre clientul BOINC al utilizatorului si serverele Rosetta @ home de la Universitatea din Washington; HTTPS (portul 443) este utilizat in timpul schimbului de parole.
Controlul de la distanta si local al clientului BOINC utilizeaza portul 31416 si portul 1043, care ar putea fi deblocat in mod specific, daca se afla in spatele unui firewall.
Unitatile de lucru care contin date despre proteine individuale sunt distribuite de la serverele localizate in laboratorul Baker de la Universitatea Washington la calculatoarele voluntarilor, care apoi calculeaza o predictie a structurii pentru proteina alocata. Pentru a evita predictiile structurii duplicat pe o proteina data, fiecare unitate de lucru este initializata cu un numar de input-uri aleator.
Aceasta ofera fiecarei predictii o traiectorie unica de descendenta de-a lungul peisajului energetic al proteinei. Predictiile structurii proteice de la Rosetta @ home sunt aproximatii ale unui minim global in peisajul energetic al unei proteine date. Acest minim global reprezinta conformatia cea mai favorabila din punct de vedere energetic a proteinei, adica starea sa naturala.
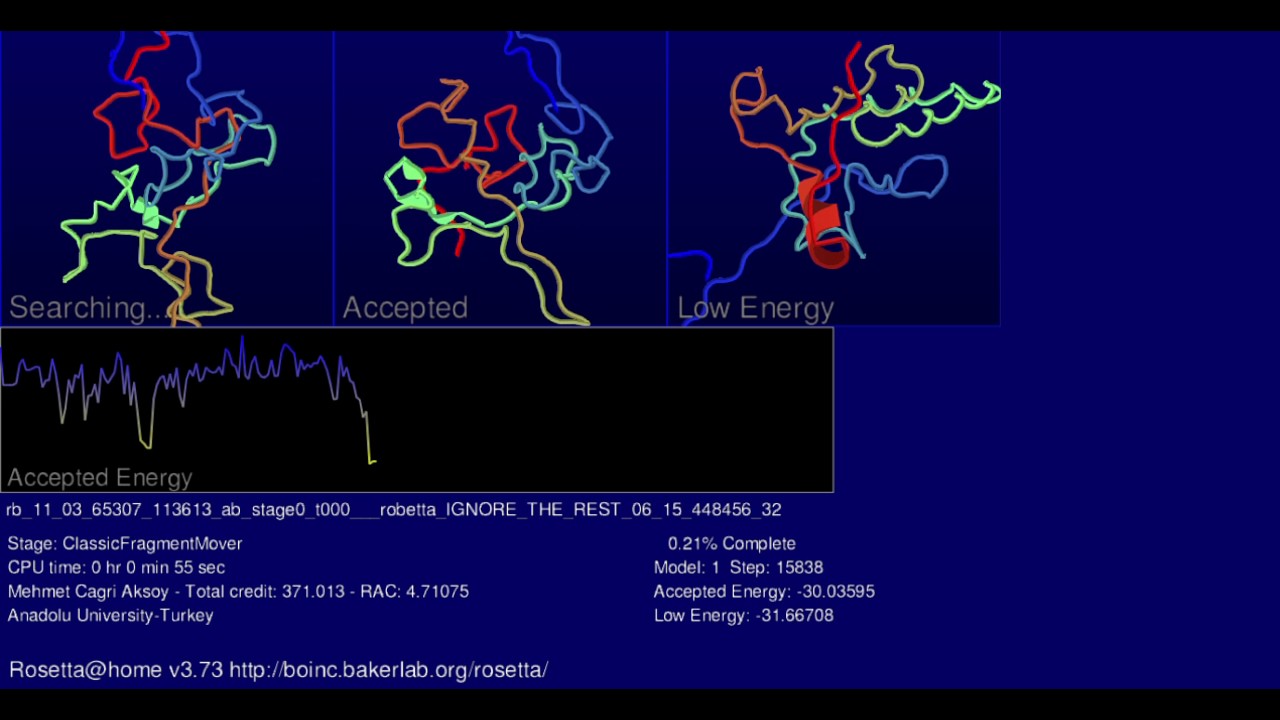
O caracteristica principala a interfetei cu utilizatorul grafic Rosetta @ home (GUI) este un screensaver care arata progresul unui workunit curent in timpul procesului de pliere a proteinelor simulate. In partea stanga sus a protectorului de ecran actual, proteina tinta este aratata adoptand diferite forme (conformatii) in cautarea sa pentru cea mai scazuta structura energetica.
Infatisata imediat in dreapta este structura celor mai recent acceptate. In partea dreapta sus este afisata conformatia energetica cea mai mica a decorului actual; sub aceasta se afla adevarata structura a proteinei, daca aceasta a fost deja determinata. Trei grafice sunt incluse in screensaver. Aproape de mijloc, este afisat un grafic pentru energia libera termodinamica a modelului acceptat, care fluctueaza pe masura ce modelul acceptat se schimba.
Un grafic al deviatiei radacina-medie-patrat (RMSD) a modelului acceptat, care masoara cat de asemanator structural este modelul acceptat cu modelul autohton, este aratat la dreapta. In dreapta graficului de energie acceptat si sub graficul RMSD, rezultatele acestor doua functii sunt utilizate pentru a produce o diagrama de energie vs. RMSD, deoarece modelul este perfectionat progresiv [12].
Ca toate proiectele BOINC, Rosetta @ home ruleaza in fundalul computerului utilizatorului, folosind o sursa de alimentare inactiva a computerului, fie la sau inainte de logare intr-un cont pe sistemul de operare gazda. Programul elibereaza resurse de la procesor, asa cum sunt necesare altor aplicatii, astfel incat utilizarea normala a computerului nu este afectata.
Multe setari ale programului pot fi specificate prin preferintele contului utilizatorului, inclusiv: procentul maxim de resurse de procesare pe care programul il poate utiliza (pentru a controla consumul de energie electrica sau producerea de caldura de pe un computer care functioneaza la o capacitate sustinuta), orele din zi in care programul poate rula. , si multe altele.
Rosetta, software-ul care ruleaza pe reteaua Rosetta @ home, a fost rescris in C ++ pentru a permite o dezvoltare mai usoara decat cea permisa de versiunea sa originala, care a fost scrisa in Fortran. Aceasta noua versiune este orientata spre obiect si a fost lansata pe 8 februarie 2008. Dezvoltarea codului Rosetta este facuta de Rosetta Commons. Software-ul este autorizat gratuit pentru comunitati academice si este disponibil pentru companiile farmaceutice contra cost.
Semnificatia/importanta proiectului
Odata cu proliferarea proiectelor de secventiere a genomului, oamenii de stiinta pot deduce secventa de aminoacizi sau structura primara a multor proteine care indeplinesc functii in interiorul celulei. Pentru a intelege mai bine functia si protejarea unei proteine in proiectarea rationala a medicamentului, oamenii de stiinta trebuie sa cunoasca structura tertiara tridimensionala a proteinei.
Structurile 3D de proteine sunt determinate in prezent experimental prin cristalografie cu raze X sau prin spectroscopie magnetica nucleara (RMN). Procesul este lent (poate dura saptamani sau chiar luni pentru a afla cum se cristalizeaza o proteina pentru prima data) si costisitor (in jur de 100.000 USD per proteina).
Din pacate, ritmul in care sunt descoperite secvente noi depaseste cu mult rata de determinare a structurii – din mai mult de 7.400.000 de secvente proteice disponibile in baza de date proteica neredundanta (nr) a Centrului National pentru Informatii Biotehnologice (NCBI), mai putin de 52.000 de structuri 3D de proteine au fost rezolvate si depuse in Banca de date proteice, principalul depozit de informatii structurale despre proteine.
Unul dintre obiectivele principale ale Rosetta @ home este de a prezice structuri proteice cu aceeasi precizie ca metodele existente, dar intr-un mod care necesita mult mai putin timp si bani.
Rosetta @ home dezvolta, de asemenea, metode pentru a determina structura si andocarea proteinelor de membrana (de exemplu, receptorii cuplati de proteine G (GPCR), care sunt exceptional de dificil de analizat cu tehnici traditionale precum cristalografia cu raze X si spectroscopia RMN, reprezinta majoritatea tintelor pentru medicamentele moderne.
Rosetta @ home este de asemenea utilizat in predictia de andocare a proteinei, care determina structura proteinelor complexe multiple sau structura cuaternara. Acest tip de interactiune proteica afecteaza multe functii celulare, incluzand legarea antigenului-anticorp si inhibitorii enzimei si importul si exportul celular.
Determinarea acestor interactiuni este esentiala pentru proiectarea medicamentelor. Rosetta este folosita in experimentul de evaluare critica a prezicerii interactiunilor (CAPRI), care evalueaza starea campului de andocare a proteinelor similar cu modul in care progresele CASP progreseaza in predictia structurii proteice.
Puterea de calcul pusa la dispozitie de voluntarii proiectului Rosetta @ home a fost citata ca un factor major in performanta Rosetta in CAPRI, unde predictiile sale de andocare au fost printre cele mai precise si complete.








